Neues Verfahren für die Abstandsmessung innerhalb von Zellen
Wissenschaftlerinnen und Wissenschaftler der Universitäten Konstanz und Bielefeld sowie der ETH Zürich weisen zum ersten Mal nach, dass mithilfe des gepulsten Elektronenspinresonanz-Spektroskopieverfahrens RIDME (relaxation-induced dipolar modulation enhancement) Abstände in Biomakromolekülen innerhalb von Zellen gemessen werden können. Dabei zeigt RIDME deutliche Vorteile gegenüber dem herkömmlichen Doppel-Elektronen-Elektronen-Resonanz-Verfahren (DEER).
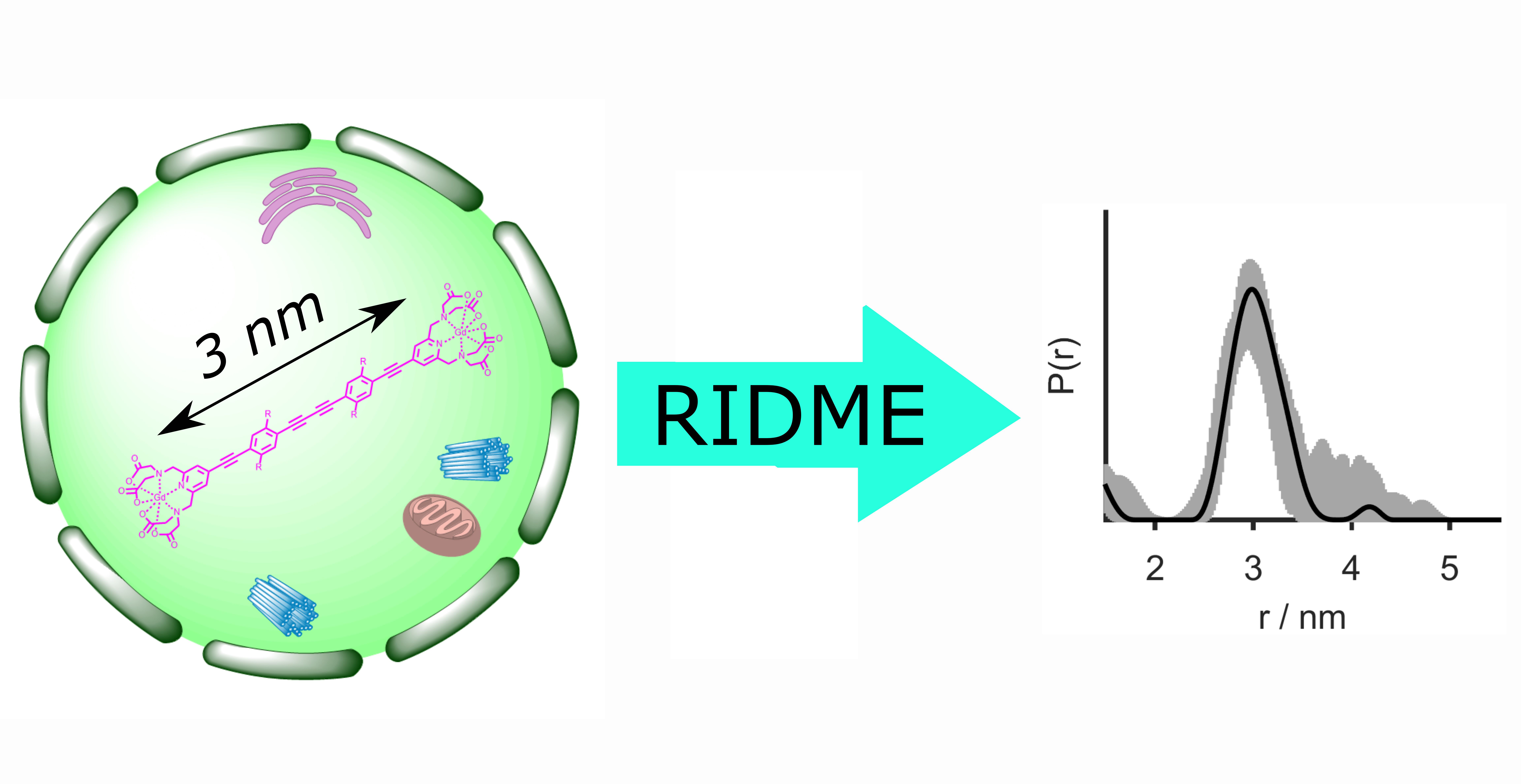
In einer gemeinsamen Veröffentlichung im Journal of Physical Chemistry Letters weisen Wissenschaftlerinnen und Wissenschaftler der Universitäten Konstanz und Bielefeld sowie der ETH Zürich zum ersten Mal nach, dass das Elektronenspinresonanzverfahren RIDME (relaxation-induced dipolar modulation enhancement) für die Messung von Abständen zwischen Gadolinium(III)-basierten Spinsonden innerhalb von Zellen genutzt werden kann. Die Abstandsmessung innerhalb von Zellen mithilfe der Elektronenspinresonanz-Spektroskopie (ESR) liefert wichtige strukturelle Informationen über Biomakromoleküle, von ihrer Konformation – der räumlichen Anordnung ihrer drehbaren Bindungen – bis hin zur Faltung und Entfaltung.
Indem es sich relaxations-induzierte Spinflips zunutze macht, um den Abstand zwischen zwei ungepaarten Elektronen oder Spinsonden zu ermitteln, überwindet das Einfrequenz-Verfahren RIDME die Nachteile bisher genutzter Techniken wie das Doppel-Elektronen-Elektronen-Resonanz-Verfahren (DEER). Damit bietet RIDME eine erhöhte Sensitivität, liefert eine bis zu fünf Mal größere Modulationstiefe und ist technisch einfacher umsetzbar.
Das Besondere daran ist, dass es den Wissenschaftlern erlaubt, unter nativen Bedingungen mit Molekülen zu arbeiten, wie Prof. Dr. Malte Drescher und Erstautor Dr. Mykhailo Azarkh von der Universität Konstanz betonen: „Unser Ausgangspunkt war, die Konformation eines Proteins in der Zelle zu analysieren. Bei Verfahren mit geringerer Sensitivität müssen wir eine große Menge Protein in die Zelle einbringen und markieren, um es beobachten zu können. Das ist natürlich nicht das, was in der Natur passiert. Idealerweise möchten wir mit Konzentrationen arbeiten, die physiologisch relevant sind. Da RIDME eine wesentlich höhere Sensitivität als DEER liefert, können wir genau das tun. Angewandt auf das Innere der Zelle versetzt uns das Verfahren in die Lage, Fragen zu beantworten, die wir sonst nicht beantworten könnten.“
Der Einsatz des RIDME-Verfahrens innerhalb der Zelle wurde im Q-Band unter Nutzung eines starren, mit Gd(III)-PyMTA markierten molekularen Lineals getestet, das in die Eizellen des Xenopus laevis (Afrikanischer Krallenfrosch) eingebracht wurde. Die Forscherinnen und Forscher nutzten damit ein Modellsystem, bei dem die genauen Abstände zwischen den Spinsonden bereits bekannt waren, was die Verifikation der mit dem RIDME-Verfahren erzielten Messergebnisse erleichtert. Die Studie mit dem Titel „Gd(III)–Gd(III) Relaxation-Induced Dipolar Modulation Enhancement for In-Cell Electron Paramagnetic Resonance Distance Determination” wurde am 13.03.2019 online im Journal of Physical Chemistry Letters veröffentlicht.
Die Nutzung von RIDME innerhalb der Zelle wurde im Rahmen des aktuellen, vom European Research Council (ERC) geförderten Projektes „SPICE – Spectroscopy in cells“ entwickelt und getestet. Für dieses Projekt wurden Malte Drescher, der die Heisenberg-Professur für „Spektroskopie komplexer Systeme“ an der Universität Konstanz innehat, und seine Arbeitsgruppe 2017 mit einem ERC Consolidator Grant in Höhe von etwa zwei Millionen Euro ausgezeichnet. Ziel ist die Entwicklung neuer spektroskopischer Ansätze zur Erforschung größerer und komplexerer biologischer Strukturen auf molekularer Ebene direkt in der Zelle.
Aufbauend auf den Ergebnissen der jüngsten Studie werden die Wissenschaftlerinnen und Wissenschaftler nun weitere geeignete Spinsonden identifizieren und RIDME so weiterentwickeln, dass es auch für die Abstandsmessung innerhalb von Molekülen eingesetzt werden kann, bei denen der Abstand zwischen den Spinsonden unbekannt ist. Ein besonderes Augenmerk wird dabei auf Molekülen liegen, die in Verbindung mit neurodegenerativen Erkrankungen wie Parkinson oder Alzheimer gebracht werden.
Faktenübersicht:
- Originalveröffentlichung: Mykhailo Azarkh, Anna Bieber, Mian Qi, Jörg W. A. Fischer, Maxim Yulikov, Adelheid Godt, Malte Drescher. Gd(III)–Gd(III) Relaxation-Induced Dipolar Modulation Enhancement for In-Cell Electron Paramagnetic Resonance Distance Determination. J. Phys. Chem. Lett. 2019, 10, pp 1477–1481. DOI: https://doi.org/10.1021/acs.jpclett.9b00340.
- Wissenschaftlerinnen und Wissenschaftler der Universitäten Konstanz und Bielefeld sowie der ETH Zürich haben zum ersten Mal nachgewiesen, dass das Elektronenspinresonanzverfahren RIDME (relaxation-induced dipolar modulation enhancement) für die Messung von Abständen innerhalb von Biomakromolekülen genutzt werden kann.
- Im Gegensatz zu anderen Verfahren wie dem Doppel-Elektronen-Elektronen-Resonanz-Verfahren (DEER) ist RIDME technisch weniger anspruchsvoll, unterliegt keinen Einschränkungen bei der Anregungsbandbreite und bietet eine bis zu fünfmal größere Modulationstiefe. Es entstehen keine Artefakte durch pseudosekulare Terme.
- Die Ergebnisse der jüngsten Studie wurden im Rahmen des aktuellen, vom European Research Council (ERC) geförderten Projektes „SPICE – Spectroscopy in cells“ erzielt. Prof. Dr. Malte Drescher, Inhaber der Heisenberg-Professur für „Spektroskopie komplexer Systeme“ an der Universität Konstanz, und seine Arbeitsgruppe wurden dafür 2017 mit einem ERC Consolidator Grant in Höhe von etwa zwei Millionen Euro ausgezeichnet. Die Projektlaufzeit beträgt fünf Jahre (2018-2022).
Presseinformation: Nr. 20/2019
