Neues Spinmarkierungsverfahren
Forscherinnen und Forscher der Universität Konstanz entwickeln ein neues Verfahren zur ortsspezifischen Spinmarkierung (Site-Directed Spin Labelling, SDSL). Es basiert auf genetisch kodierten nichtkanonischen Aminosäuren, die sich für die Diels-Alder-Chemie eignen, sowie auf einem neuen Spin Label, PaNDA.
In Kombination mit Elektronenspinresonanz-Spektroskopie (ESR) gehört die ortsspezifische Spinmarkierung (Site-Directed Spin Labelling, SDSL) zu den etablierten und leistungsstarken Verfahren für die Aufklärung von Struktur, Funktion und Dynamik von Proteinen und Proteinkomplexen. Bevorzugt werden dabei Nitroxid-basierte Spinmarker verwendet. Sie sind klein, haben keinen störenden Einfluss auf die Proteinstruktur und besitzen hervorragende spektroskopische Eigenschaften. “Das ideale Spinmarkierungsverfahren weist hohe Reaktionsraten bei hoher Selektivität auf”, erklärt Prof. Dr. Malte Drescher, Professor für die Spektroskopie komplexer Systeme am Fachbereich Chemie der Universität Konstanz und mit Prof. Dr. Valentin Wittmann, einem Experten für organische Synthese, Hauptautor der Studie.
„Allerdings kann es schwierig werden, hohe Reaktivität bei gleichzeitig hoher Selektivität zu erreichen“, so Drescher weiter. „Konventionelle Spinmarker, die beispielsweise auf Gadolinium(III) oder Trityl basieren, weisen außerdem entweder sehr große Bandbreiten bei niedrigen Modulationstiefen oder andererseits sehr schmale Bandbreiten auf, die sich für die Experimente, die wir durchführen möchten, nicht eignen.“ Eine neue Studie von Drescher, Wittmann und ihren Arbeitsgruppen vom Fachbereich Chemie der Universität Konstanz, die am 14. August 2019 online im Journal ChemBioChem veröffentlicht wurde, stellt ein neuartiges Spinmarkierungsverfahren vor. Im Gegensatz zu den herkömmlichen Verfahren hebt es sich durch Nitroxid-basierte Spinmarker und genetisch codierte nichtkanonische Aminosäuren als Angriffspunkt für SDSL hervor.
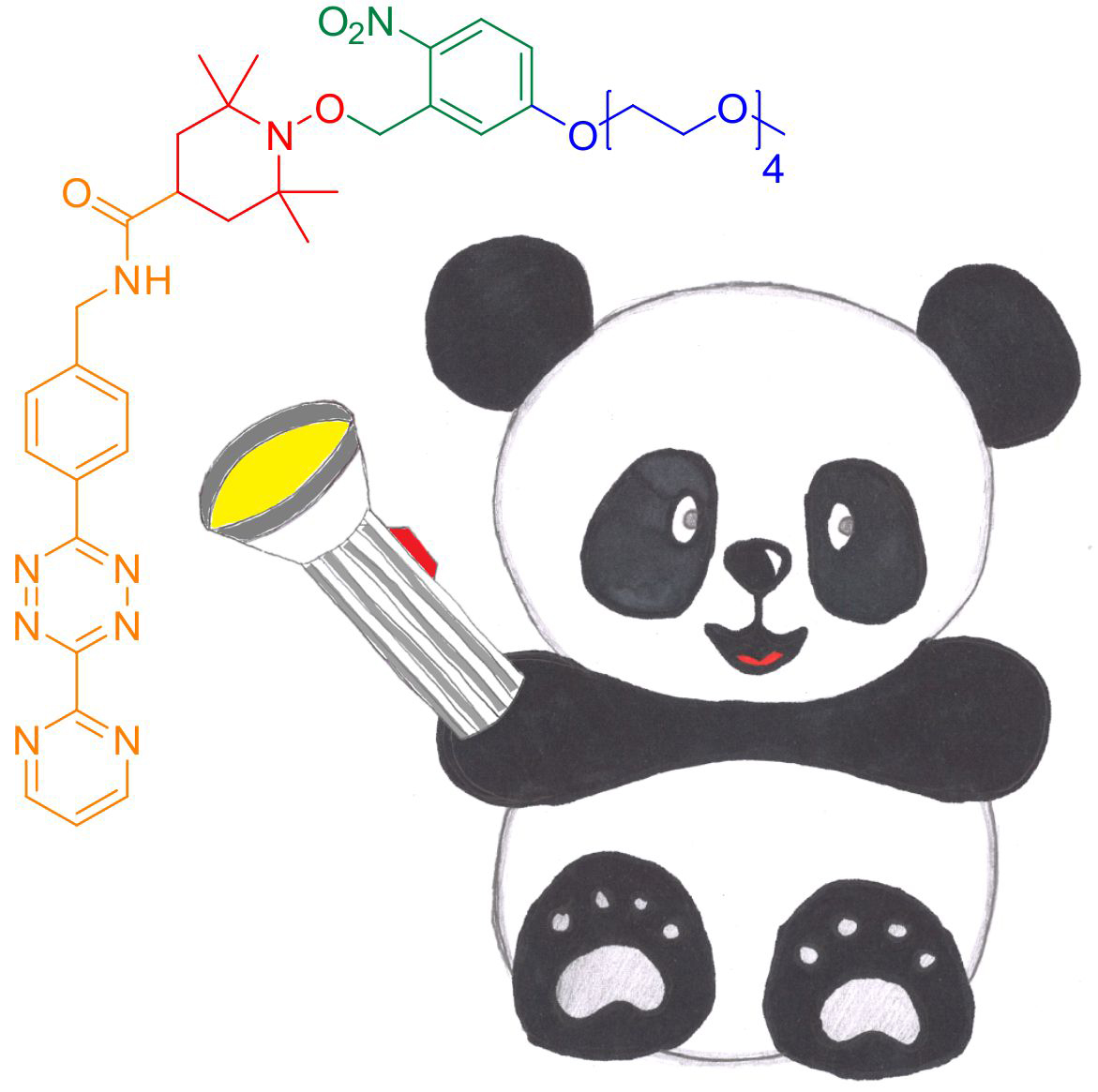
„Durch die Nutzung von Nitroxiden erhalten wir ideale Bandbreiten und den Zugang zu dynamischen Informationen“, erklärt dazu Anandi Kugele, Doktorandin an der Konstanzer Graduiertenschule Chemische Biologie und Erstautorin des Papers. Für die Vorstellung der darin beschriebenen neuen Forschungsergebnisse bei der diesjährigen Ausgabe der Rocky Mountain Conference on Magnetic Resonance in Denver, Colorado (USA) erhielt sie jüngst ein renommiertes Reisestipendium des National High Magnetic Field Laboratory in Tallahassee, Florida (USA). „Herkömmliche Nitroxid-basierte Marker haben eine begrenzte Redox-Stabilität, was ihre Nutzung in der Zelle erschwert. Die Herausforderung für uns bestand darin, die Stabilität des Nitroxids zu erhöhen und Nitroxid-basierte Spinmarker so für zukünftige Routineeinsätze bei in vivo-Experimenten anzupassen.“ Die Forscher entwickelten daher einen neuen Spinmarker, der mittels einer Diels-Alder-Cycloaddition mit inversem Elektronenbedarf (DAinv) an genetisch codierte nichtkanonische Aminosäuren in einem Protein bindet, eine Methode, die sich bereits in vielen Anwendungsbereichen sowohl in vitro als auch in vivo bewährt hat. Um das Nitroxid zu stabilisieren, nutzten die Wissenschaftlerinnen und Wissenschaftler ferner durch Licht entfernbare Schutzgruppen, um Nitroxide zunächst zu schützen und sie je nach Bedarf freizugeben.
Der neue Spinmarker mit dem Namen photoaktivierbares Nitroxid für die Diels-Alder-Reaktion mit inversem Elektronenbedarf (DAinv) (photoactivatable nitroxide for DAinv reaction, kurz PaNDA), ist wasserlöslich, für die ESR-Spektroskopie geeignet und lässt sich effektiv entschützen, wie sowohl in vitro- als auch Lysat-Tests mit den beiden Modell-Proteinen grün fluoreszierendes Protein (GFP) und Escherichia coli Thioredoxin (TRX), das in fast allen bekannten Organismen vorkommt, vermuten lassen. „Unsere nächste Aufgabe wird sein, an dem Verfahren zu feilen, mit dem wir den PaNDA Spinmarker in das Innere von Zellen transportieren. Außerdem wollen wir unter anderem testen, wie effizient die Markierung und die Entschützung in der Zelle ablaufen“, resümiert Malte Drescher: „Unsere Forschungsergebnisse zeigen jedoch klar, dass das PaNDA-Label sich grundsätzlich für die ESR-Spektroskopie in herausfordernden Umgebungen eignet, insbesondere auch für die Anwendung im Inneren von Zellen. Unsere Tests mit E. coli-Lysat sind in dieser Hinsicht besonders vielversprechend. Langfristig könnte unser Verfahren neue Möglichkeiten zur Erforschung von Proteinen mittels ESR-Spektroskopie eröffnen.“
Faktenübersicht:
- Forscherinnen und Forscher der Universität Konstanz entwickeln ein innovatives Verfahren der ortsspezifischen Spinmarkierung (Site-Directed Spin Labelling, SDSL), das auf genetisch codierten nichtkanonischen Aminosäuren, die sich für die Diels-Alder-Cycloaddition mit inversem Elektronenbedarf (DAinv) eignen, sowie einem neuen Spinmarker, PaNDA, basiert
- Das Verfahren zeigt hohe Reaktionsraten bei hoher Selektivität und bietet die Möglichkeit, das Nitroxid in Escherichia coli-Lysat zu entschützen
- Originalveröffentlichung: Anandi Kugele, Bjarne Silkenath, Jakob Langer, Valentin Wittmann, Malte Drescher, Protein Spin Labeling with a Photocaged Nitroxide Using Diels-Alder Chemistry, ChemBioChem, online veröffentlicht am 14 August 2019, DOI: https://doi.org/10.1002/cbic.201900318
- Gefördert vom Europäischen Forschungsrat (ERC) im Rahmen des Horizon 2020 Research and Innovation Programme der Europäischen Union sowie von der Deutschen Forschungsgemeinschaft (DFG, Sonderforschungsbereich 969 “Chemical and Biological Principles of Cellular Proteostasis”)
